eFLASH: Revolucionando a Imunomarcação em Tecido Inteiro com Processamento Uniforme
Legenda: a. Efeito do pH e da concentração de NaDC na ligação dos anticorpos anti-NPY e anti-PV. Intensidades médias normalizadas de fluorescência do soma (n = 3 experimentos independentes para cada condição de tampão, com cada soma representada como um ponto de dados individual; soma total segmentada: NPY = 160, PV = 256). Comparações múltiplas ANOVA unidirecional, média ± desvio padrão (s.d.). ****P < 0,0001; ***P = 0,0003 (anti-PV 1% NaDC, pH 7,5 versus pH 10); ***P = 0,0004 (anti-NPY pH 7,5, 0% NaDC versus 0,1% NaDC); *P = 0,0243 (anti-NPY 1% NaDC, pH 7,5 versus pH 10).
b. Imagens representativas dos dados em a, exibidas em dois intervalos de exibição diferentes: 0/4.095 (linha superior) e 0/1.500 (linha inferior). Barra de escala: 20 µm (branco).
c. Esquema do sistema eFLASH. O pH e a concentração de NaDC da solução de marcação são reduzidos gradualmente para ajustar a afinidade de ligação das sondas moleculares de baixa para normal no contexto de SE. A oxidação eletrocatalítica do d-sorbitol na superfície do ânodo gera componentes ácidos que reduzem o pH. A concentração de NaDC na solução de marcação é reduzida pela difusão de monômeros de NaDC através da membrana nanoporosa.
d. Medição do pH (n = 4 experimentos independentes) e da concentração de NaDC (n = 6 experimentos independentes) durante o processamento de 24 horas. Dados representados como média ± erro padrão da média (s.e.m.).
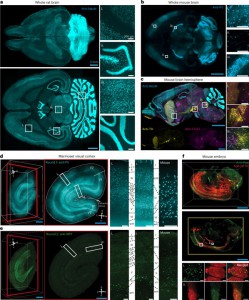
e, f. Marcação volumétrica de dois hemisférios de um único cérebro utilizando SE (e) e eFLASH (f), com a mesma massa de anticorpos: 3 µg de anti-CB (calbindina) e 5 µg de anti-NF (marcador de neurofilamento). Planos ópticos de dados volumétricos 3D exibidos como projeções de intensidade máxima (MIPs) de 20 µm. Visualizações sagital (plano de imagem original) e coronal (plano reconstruído). As distâncias dos planos sagitais ópticos em relação ao plano medial são z = 2,8 mm (e) e z = 3,1 mm (f).
Visualizações ampliadas:
Camadas coronais: e-i e f-i
Estriado: e-ii e f-ii
Cerebelo: e-iii e f-iii
Barras de escala: 2 mm (ciano) e 100 µm (branco). n = 2 experimentos independentes. MIP: projeção de intensidade máxima
Creditos da imagem: Yun et al, 2025
Este estudo apresenta uma inovação tecnológica que resolve desafios fundamentais na análise molecular em tecidos integrais: o método Continuous Redispersion of Volumetric Equilibrium (CuRVE), implementado no sistema **eFLASH (electrophoretic-Fast Labeling using Affinity Sweeping in Hydrogel)**. Essa abordagem permite a imunomarcação volumétrica rápida, uniforme e de alta resolução em órgãos inteiros, como cérebros de camundongos e ratos, além de blocos de tecido humano, em apenas 24 horas.
O CuRVE aborda a limitação do transporte desigual de anticorpos e reagentes em tecidos densos, ajustando lentamente o ambiente químico ao longo do tempo para manter o equilíbrio espacial uniforme. No eFLASH, isso é alcançado combinando transporte eletroforético e varredura gradual de condições químicas, como pH e concentração de detergentes (ácido desoxicólico). Essa metodologia garante que todas as células em tecidos intactos experimentem o mesmo processamento químico, independentemente de sua localização.
Os resultados demonstraram a capacidade do eFLASH de rotular múltiplos marcadores moleculares simultaneamente, como NeuN, PV e ChAT, em diferentes tecidos e espécies (incluindo primatas não humanos e organoides humanos), sem necessidade de otimizações extensivas. A técnica também revelou zonas de baixa expressão de parvalbumina (PV) em cérebros de camundongos saudáveis, uma descoberta que desafia preconcepções sobre a expressão celular e não foi detectada por abordagens genéticas, como modelos transgênicos baseados em CRE-LoxP.
Adicionalmente, a pesquisa identificou discrepâncias entre métodos genéticos e imunológicos para rotulação celular, destacando que muitos marcadores proteicos detectados pelo eFLASH não estavam representados em modelos transgênicos. Por exemplo, grandes diferenças foram observadas na expressão de parvalbumina e colina acetiltransferase (ChAT) entre métodos genéticos e imunomarcação, enfatizando a necessidade de validação proteômica para estudos robustos.
O eFLASH provou ser uma ferramenta universal e escalável, compatível com uma ampla variedade de tecidos e sondas moleculares, incluindo proteínas estruturais, marcadores de atividade neuronal e moléculas de interesse patológico. Com suas propriedades de alta reprodutibilidade, custo-benefício e robustez contra variabilidades experimentais, o eFLASH tem o potencial de revolucionar a análise molecular espacial em larga escala, auxiliando no avanço de pesquisas transgênicas, caracterização de modelos patológicos e validação de amostras clínicas humanas.
Essa tecnologia representa um marco para estudos multiplexados e de alta resolução, abrindo caminhos para investigações detalhadas do proteoma em tecidos integrais e auxiliando na compreensão da biologia em nível celular e molecular com precisão espacial.
Uniform volumetric single-cell processing for organ-scale molecular phenotyping
eFLASH: Processamento Uniforme de Células em Escala Orgânica para Imunomarcação Avançada