Metilação gênica: qual metiltransferase controla a metilação de cada gene?
Créditos da imagem: Jensen et al, 2019
A análise gnêrmica dos padrões de metilação do DNA usando sequenciamento de DNA em tempo real aumentou muito o número de metilomas disponíveis publicamente, ou seja, já se sabe quais, e onde, diferentes genes são metilados e possuem sua expressão modulada.
No entanto, ainda não existe uma ferramenta que combina padrões de metilação e os correspondentes genes da metiltransferases (DNMTs). Dentre todas as DNMTs, quais são responsáveis por metilar quais genes?
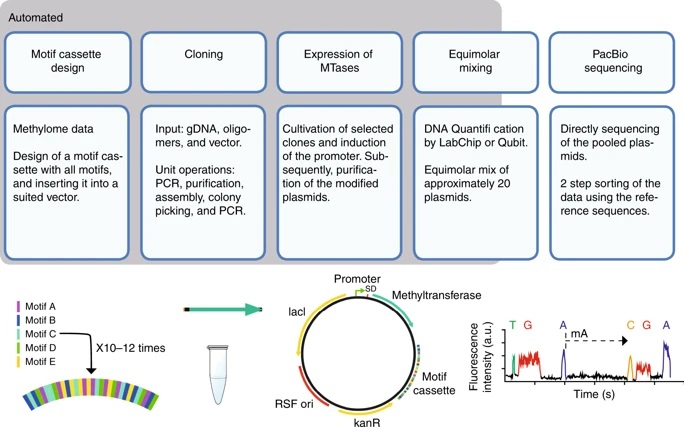
Neste trabalho, pesquisadores demonstram método de alto rendimento para o acoplamento de metiltransferases com seus respectivos motivos de metilação, usando clonagem automatizada e analisando as metiltransferases em vetores contendo um cassete específico de tensão contendo todos os potenciais locais-alvo. Para validar o método, foram avaliados os genomas dos microorganismos termófilos Moorella thermoacetica e do mesófilo Acetobacterium woodii, duas bactérias acetogênicas com genomas substancialmente modificados com 12 motivos de metilação e um total de 23 genes de metiltransferases. Utilizando este método, foram caracterizadas as 23 metiltransferases e atribuídos quais os motivos de metilação correspondem às respectivas enzimas.
Com esta técnica, será possível compreender qual o efeito da deleção de determinada DNMT na expressão de genes específicos, alvos de metilação!
Veja os detalhes deste trabalho no paper anexado :
Genome-wide systematic identification of methyltransferase recognition and modification patterns