Observando a célula em seu estado nativo: Imageando a dinâmica subcelular em organismos multicelulares.
Créditos da imagem: Tsung-Li Liu et al, 2018.
Há anos, os cientistas têm visualizado células vivas com técnicas de microscopia, mas as melhores resoluções sempre foram obtidas quando estas células estavam isoladas em uma monocamada sobre lâminulas de vidro. Grandes concentrações de células dentro de organismos inteiros difratam desorganizadamente a luz, como se iluminássemos um saco de bolinhas de gude com uma lanterna pequena. Desta maneira, visualizar células em seu estado nativo dentro de um tecido sempre foi um grande desafio
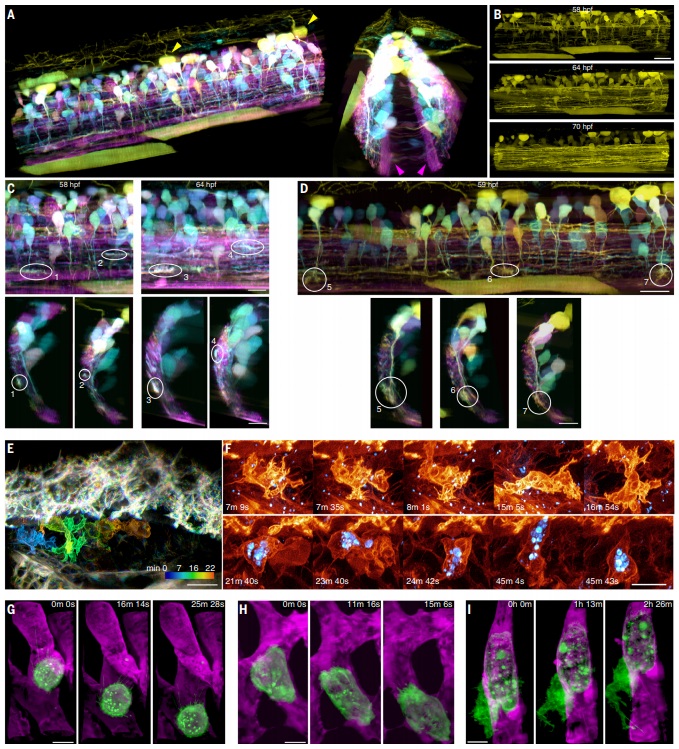
Combinando duas tecnologias de imagem, os cientistas agora podem observar em detalhes 3D sem precedentes o movimento migratório de células cancerígenas, os circuitos nervosos espinhais e as células do sistema imunológico cruzando as barreiras em direção ao ouvido interno zebrafish. Para organizar a luz emitida pelas células in situ quando iluminadas, os pesquisadores se voltaram para a óptica adaptativa – a mesma tecnologia usada pelos astrônomos para fornecer visões claras de objetos celestes distantes através da atmosfera turbulenta da Terra. Então, para criar imagens da coreografia interna dessas células de forma rápida, porém suave, em 3-D, a equipe usou lattice light sheet microscopy. Essa tecnologia varre rápida e repetidamente uma camada super fina através da célula enquanto adquire uma série de imagens em 2-D, construindo um filme 3D de alta resolução de dinâmica subcelular.
O novo microscópio é essencialmente dois microscópios em um: um sistema óptico adaptativo para manter uma fina espessura da camada celular iluminada dentro de um organismo, e outro sistema óptico adaptativo para criar imagens sem distorção ao olhar para baixo no plano iluminado a partir de um ponto superior. Seria como se observássemos a sombra da célula projetada abaixo dela quando esta é iluminada. Ao lançar um laser através de um dos caminhos, os pesquisadores criam um ponto de luz brilhante na região que desejam visualizar. Nesse caso, é possível montar grandes imagens 3-D de uma série de subvolumes, cada um com suas próprias correções independentes de excitação e detecção.
Veja este trabalho na íntegra no paper anexado:
Observing the cell in its native state- Imaging subcellular dynamics in multicellular organisms